零基础入门转录组分析——数据处理(TCGA数据库)
零基础入门转录组分析——数据处理(TCGA数据库)
目录
- 零基础入门转录组分析——数据处理(TCGA数据库)
- 1. 数据集获取
- 2. 数据处理(Rstudio)
- 配套资源链接:[配套资源(代码+原始数据+处理好的数据)](https://download.csdn.net/download/weixin_49878699/89052340)
TCGA应该是肿瘤数据最权威的来源之一,但是从TCGA上下载数据集相对来说比较麻烦,因此出现了很多针对TCGA数据进行二次开发的衍生网站,XENA.UCSC就是很直观强大的一个在线网站,里面收录了众多数据库的数据集,其中就包括了TCGA数据集,并且是整合好的,可以直接用于分析。
并且XENA.UCSC这个网站不仅能下载数据,还能进行在线分析,例如:KM分析,表达量分析等,详细情况可以参考好用的TCGA分析工具:UCSC Xena,但是在这篇教程中仅介绍如何从UCSC上下载所需要的TCGA数据集,并且下载后在R中对数据集进一步处理成后续分析所要的形式。
本项目以非小细胞肺癌(non-small cell lung cancer,NSCLC)中的肺腺癌(lung adenocarcinoma,LUAD)作为展示 选用的数据库是TCGA。 物种:人类(Homo sapiens) 实验分组:疾病组,对照组。 我这里使用的R版本是4.2.2 要用到的R包:tidyverse, rtracklayer, dplyr
1. 数据集获取
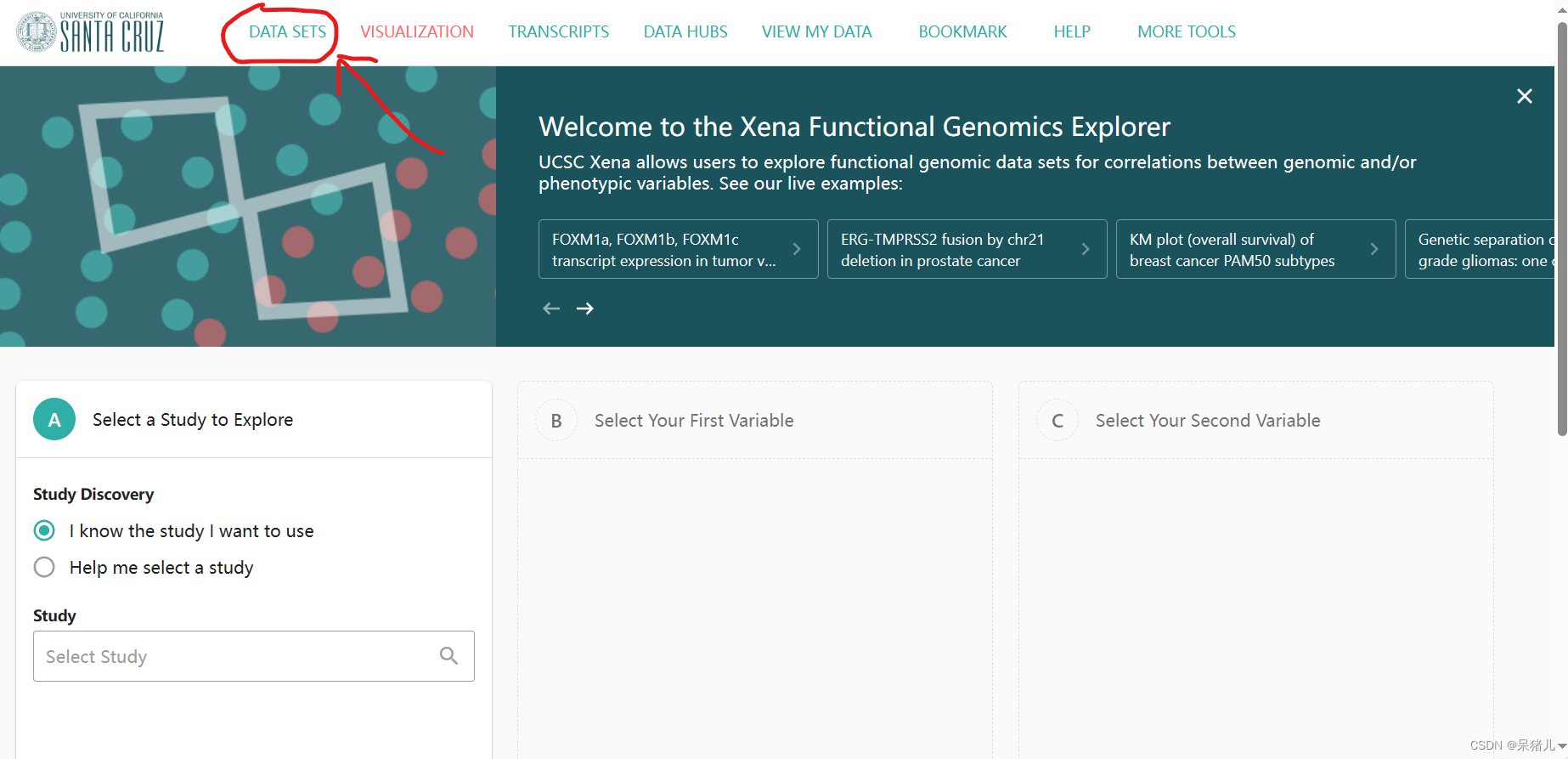
首先进入XENA.UCSC官网(如下图所示),点击箭头指向的位置进一步运行Xena。
进一步点击DATA SETS进入Xena中存储数据集的页面
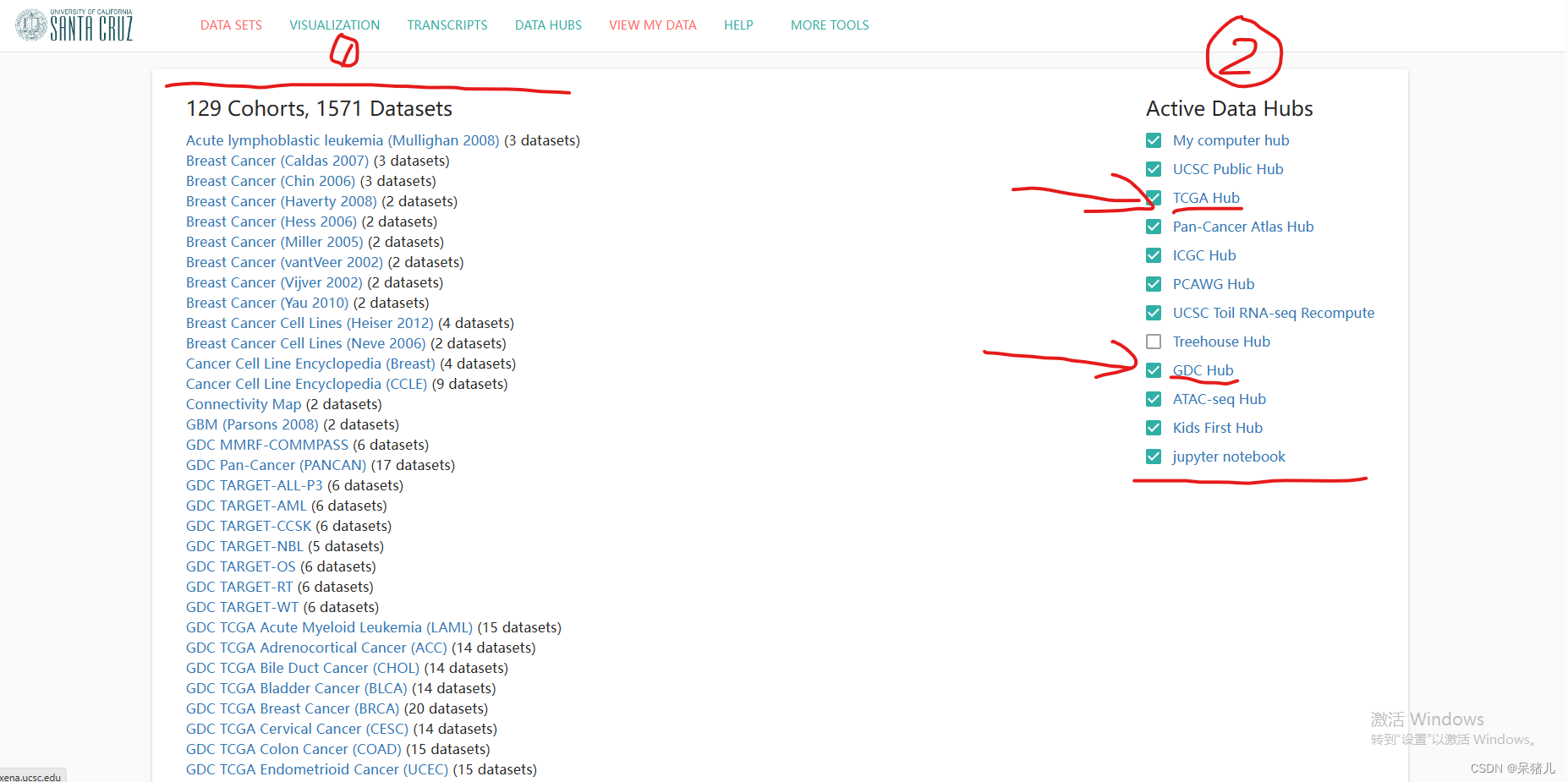
如下图所示为Xena中存储数据集的页面,其中(1)就是收录的各种疾病的数据集,(2)是筛选条件,由于该网站不仅收录了TCGA数据库的,同样还收录了其他数据库的数据集,因此可以根据自己的需要选择想要的数据库及数据集。我们想要下载是TCGA数据集,因此勾选GDC Hub 和TCGA Hub(至于为什么会选择这两个,在下面会有个人的一点理解和解释)
如下图所示,为勾选GDC Hub 和TCGA Hub的筛选情况,可以从图中看到两个都是TCGA数据集,那么区别是什么呢?
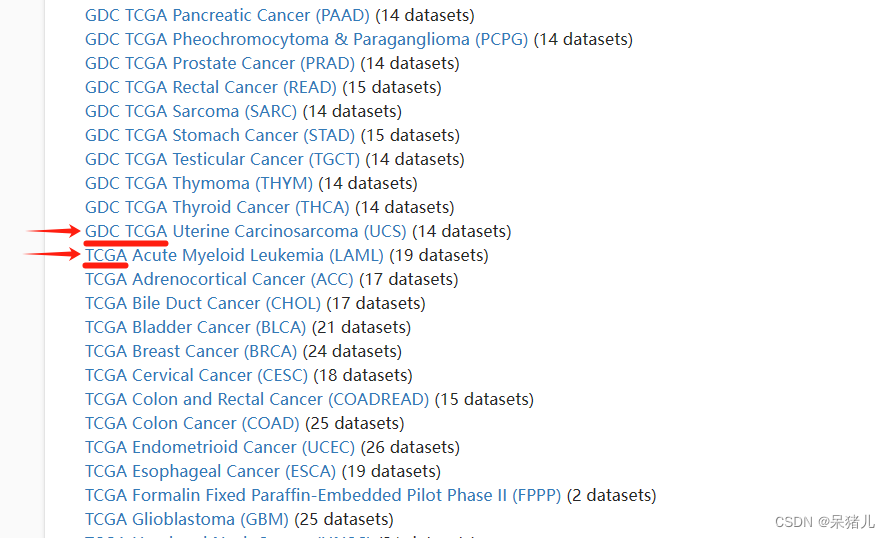
直接以本教程要展示的疾病——肺腺癌(lung adenocarcinoma,LUAD)为例,分别找到对应的GDC-TCGA数据集和TCGA数据集,如下所示,分别点进去可以看到详细信息。
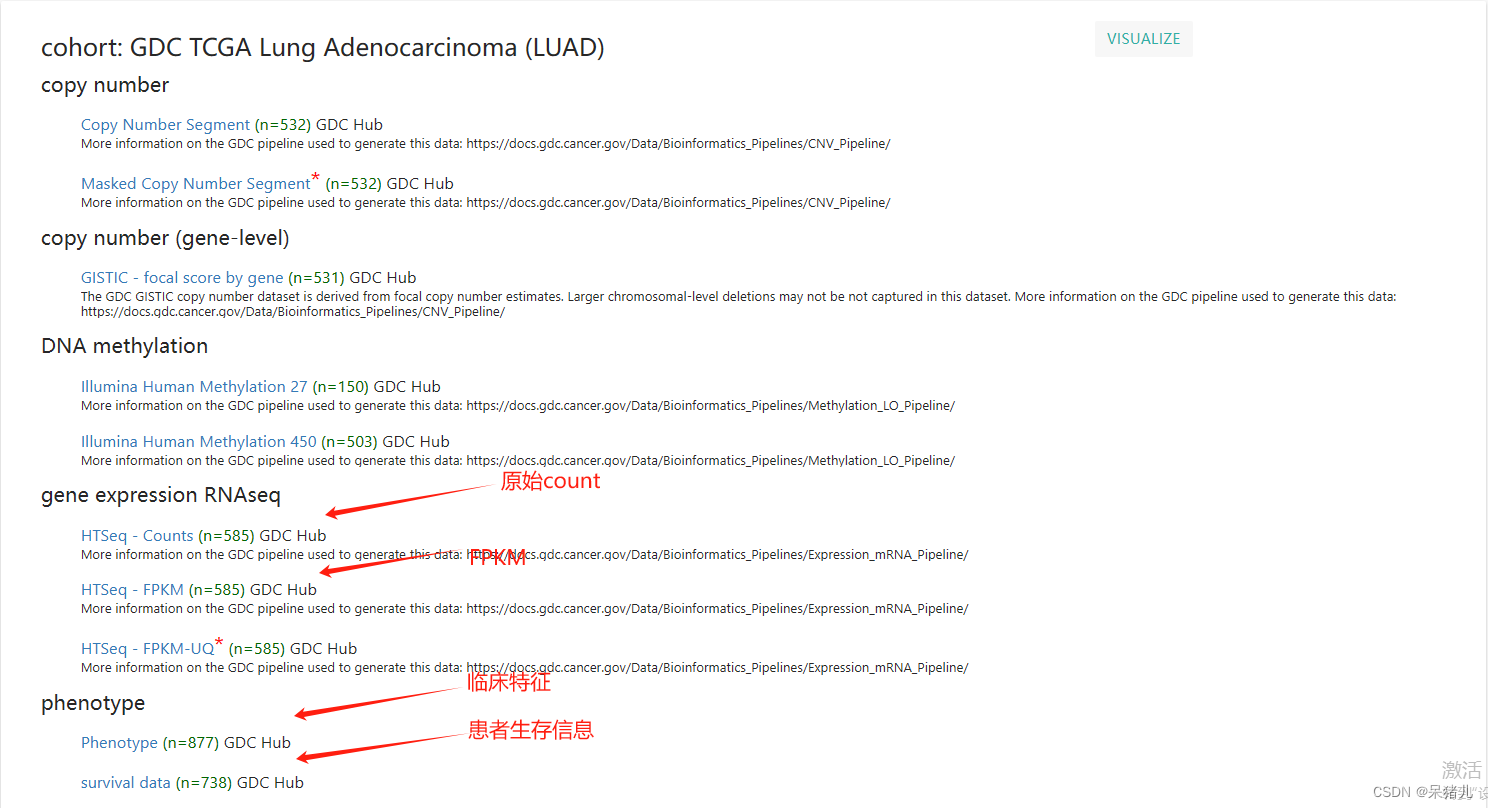
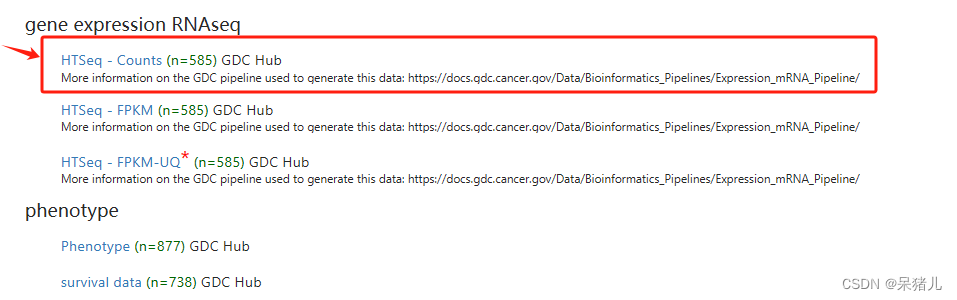
如下图所示为GDC-TCGA肺腺癌数据集,可以看到整个界面干净简洁,并且图中标注出了4个红字部分就是我们后续要用到的数据,分别是原始Count,FPKM,Phenotype,以及survival data。
4个数据解释如下:
- counts——转录组的原始表达矩阵,里面对应的基因表达量又被称作raw_count,行名为基因symbol,列名为样本名(也是病人的id,可以将每一列看作一个病人)
- fpkm——raw_count经过转换后的表达矩阵,其计算公式可参考一文了解Count、FPKM、RPKM、TPM | 相互间的转化 | 收藏教程
- phenotype——病人的临床信息,包含分组信息,肿瘤分期,分级,年龄,性别等
- survival——病人的生存信息,里面通常会有4列信息,两列的病人的id,另外两列:OS——生存状态(0表示存活,1表示死亡),OS.time——生存时间
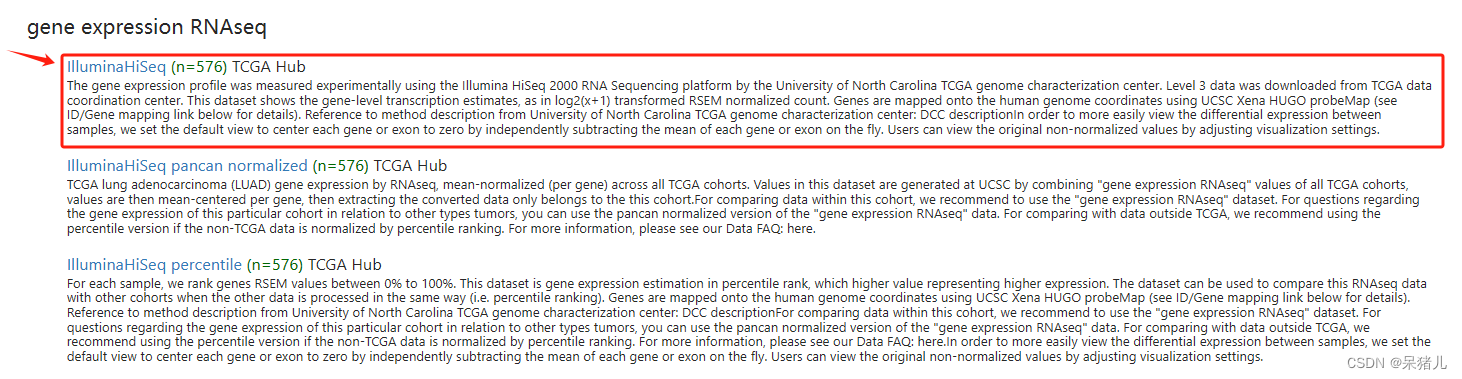
如下图所示为TCGA肺腺癌数据集,可以看到整个界面信息比较多,但也包括了基因表达量及患者的临床信息和生存信息。
那么这两者区别是什么?可参考别人的教程:UCSC Xena数据库中GDC TCGA数据和TCGA数据的区别和聊UCSC xena的数据下载问题,但是解释的都不是很清楚,并且官方解释也是晦涩难懂,对于后续分析该选择哪个并没有做出很好的解答。
总的来说就是TCGA这个数据集的发行年份更早,而GDC则是发行年份更晚一些,做科研的小伙伴也都知道一般都要追前沿,那我们这也不例外,选择一个最新的GDC数据库。
同时于偶然间发现了一件事:GDC的数据处理要比TCGA更加方便!如下图所示,分别点进去GDC中的Counts和TCGA中的IlluminaHiSeq。
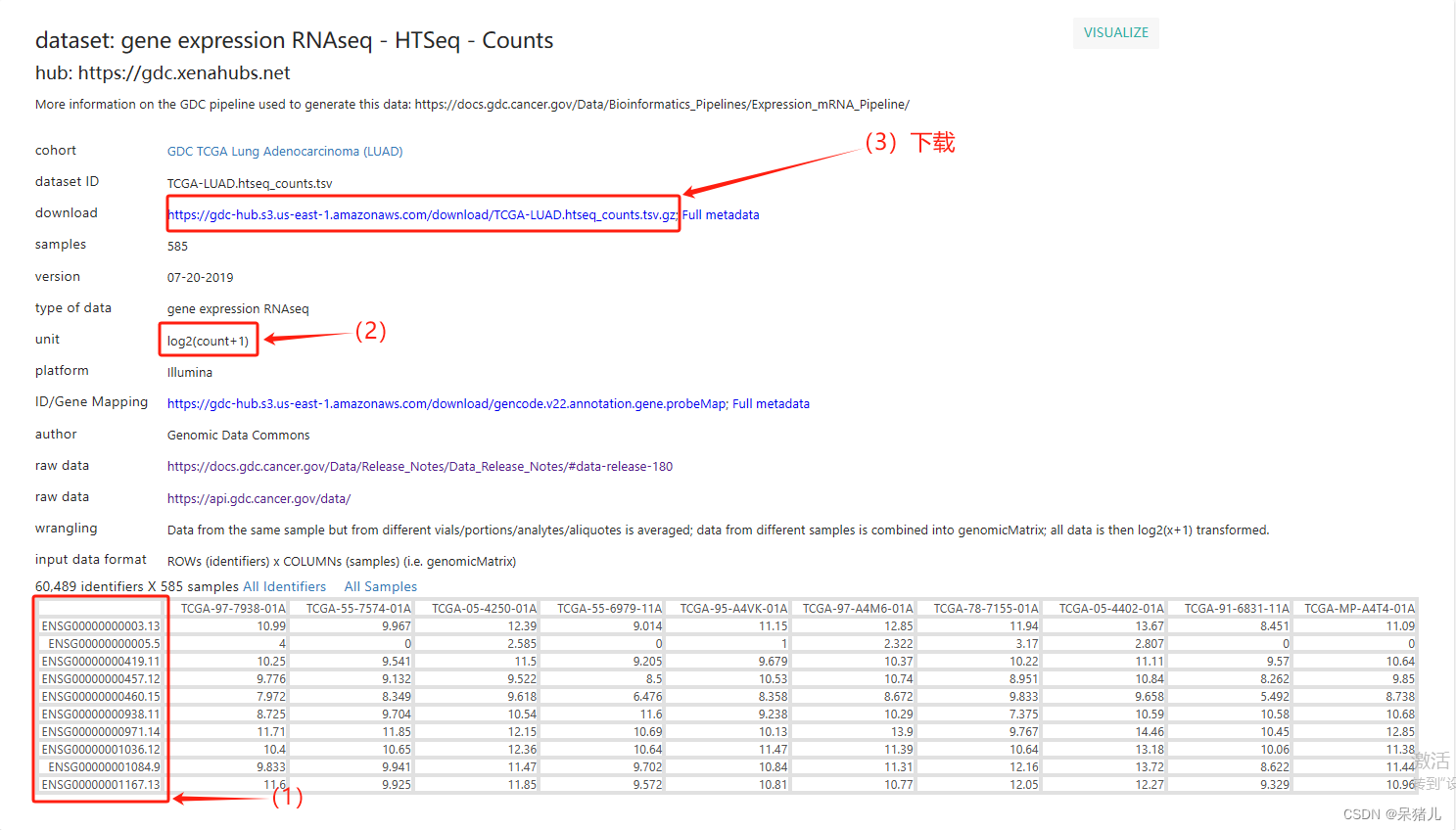
结果如下所示是GDC的数据,其中(1)是基因的ensemble_ID,这个玩意可以直接转换成基因symbol,(2)表示该数据集GDC数据库是进行了log2(X+1)处理的,因此后续处理是要反转一下才能获得想要的count数据,(3)是下载的链接,只要点击即可下载数据。
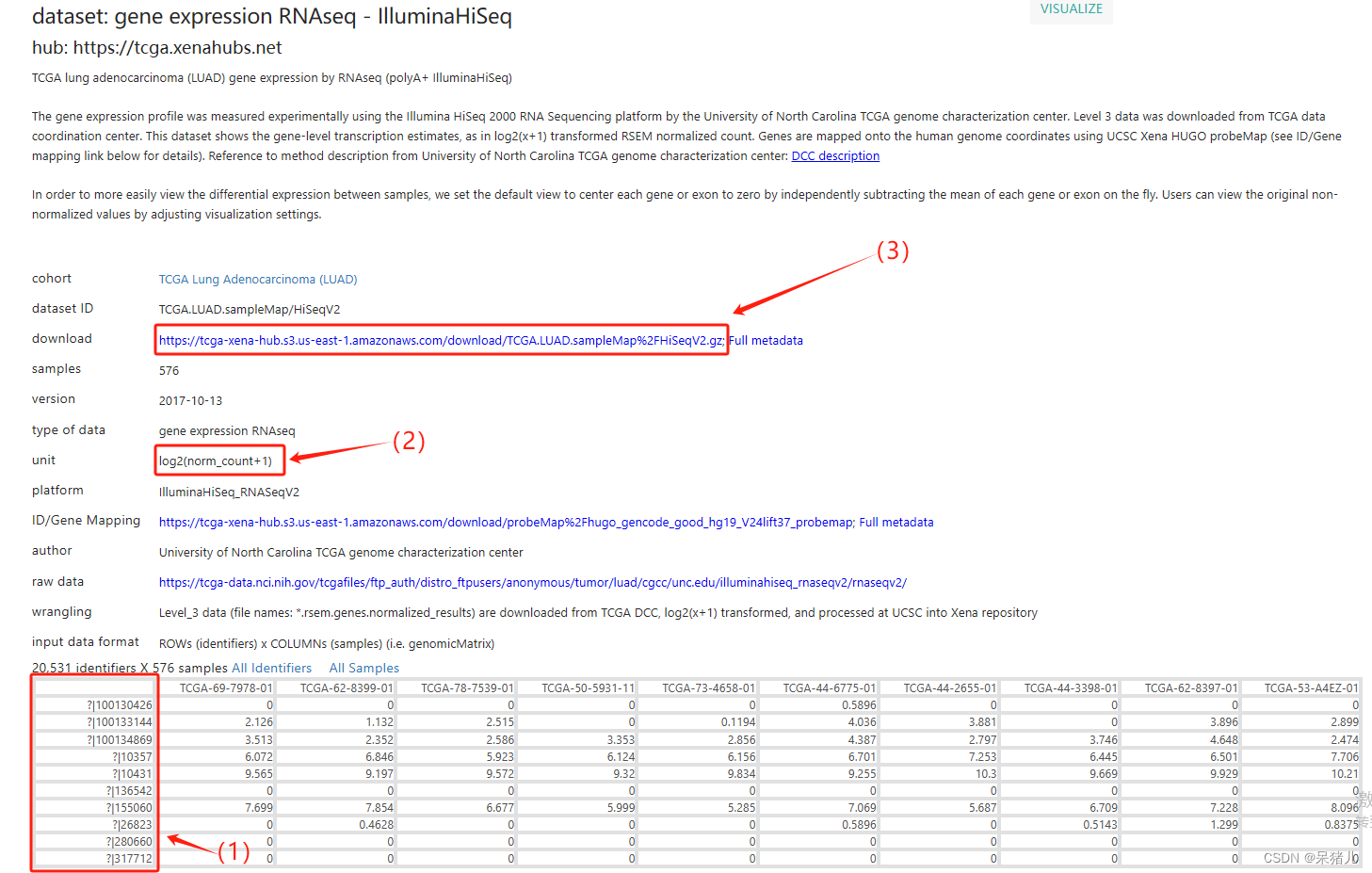
 TCGA的数据如下图所示,其中(1)对应的不知道是什么,推测应该是探针的ID(没深入研究),也有可能是发行年份较早有些基因缺失。但是这个玩意要想转换成基因symbol,应该需要先找到对应的文件,才能转换成对应基因symbol,同时细心的小伙伴也可以发现TCGA这个数据库他只有20531个基因,而前面GDC数据库则是60489个基因,(2)同样表示该数据集是进行了log2(X+1)处理的,因此后续处理也是要反转一下,(3)是下载的链接。
TCGA的数据如下图所示,其中(1)对应的不知道是什么,推测应该是探针的ID(没深入研究),也有可能是发行年份较早有些基因缺失。但是这个玩意要想转换成基因symbol,应该需要先找到对应的文件,才能转换成对应基因symbol,同时细心的小伙伴也可以发现TCGA这个数据库他只有20531个基因,而前面GDC数据库则是60489个基因,(2)同样表示该数据集是进行了log2(X+1)处理的,因此后续处理也是要反转一下,(3)是下载的链接。总的来说已目前的理解来看GDC数据库中的数据集更加方便简洁一些,可以直接通过ensemble_ID对应基因,因此选用一个更加方便的方法——GDC数据库,前面我们也说了图中(3)是下载链接;“照猫画虎,依葫芦画瓢”,以同样的方式下载FPKM,Phenotype,以及survival data,下载完后注意后缀是不是XX.tsv.gz或XX.tsv。如下图所示是下载好的数据集们,接下来要在R中让他们展示自我并做进一步处理。
2. 数据处理(Rstudio)
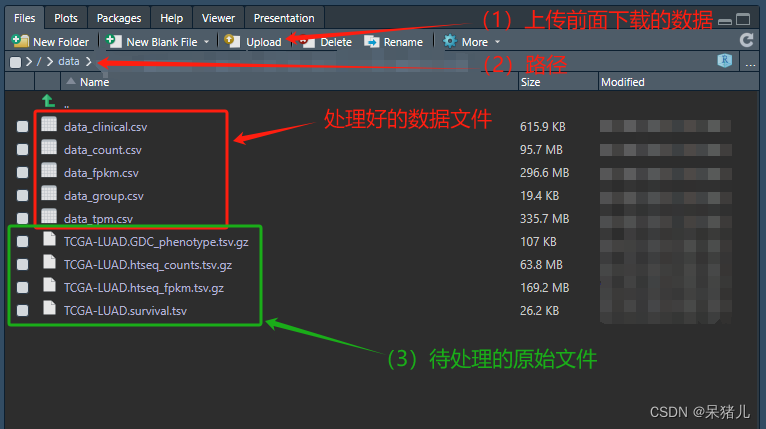
rm(list = ls()) # 删除工作空间中所有的对象 setwd('/XX/XX/XX') # 设置工作路径 if(!dir.exists('./00_rawdata')){ dir.create('./00_rawdata') } # 判断该工作路径下是否存在名为00_rawdata的文件夹,如果不存在则创建,如果存在则pass setwd('./00_rawdata/') # 设置路径到刚才新建的00_rawdata下上传前面下载的4个tsv数据到Rstudio中(注意:最好上传到刚才创建的00_rawdata文件夹下),如下图所示,绿色的框就是要处理的原始文件。
首先加载要用的包,万能的tidyverse,dplyr,rtracklayer如果没有安装这3个包,可以通过install.packages(‘XXX’)指令安装,XXX就是包的名字,例如:install.packages('tidyverse')
library(tidyverse) library(dplyr) library(rtracklayer)
### 读入下载的数据 tcga_count num mete[i, 2] = "T"} # 判断:如果提取的数字在0-9之内就在每行第二列加上T表示肿瘤样本 if(num %in% seq(10, 29)){mete[i, 2] = "N"} # 判断:如果提取的数字在10-29之内就在每行第二列加上N表示正常对照 } colnames(mete)